Hipometilación genómica global en tumores de mama canino
Resumen
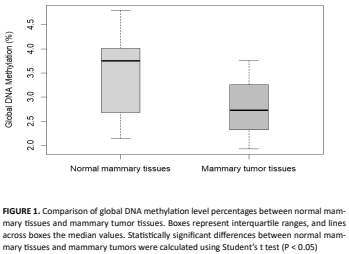
La regulación génica mediante mecanismos epigenéticos es esencial para el crecimiento y el desarrollo normal, ya que influye en el potencial transcripcional de los genes. En el cáncer de mama, las modificaciones epigenéticas desempeñan un papel clave en su desarrollo y progresión. En etapas tempranas de la carcinogénesis, como consecuencia de lesiones genéticas o factores ambientales, se producen alteraciones en la estructura de la cromatina a través de la metilación del ADN y de modificaciones postraduccionales de proteínas unidas al ADN. Al igual que varios tumores, se observa una hipometilación a nivel genómico y la hipermetilación de ciertos genes, especialmente en islas CpG que normalmente no se encuentran metiladas. Con el objetivo de comparar el nivel de metilación global del ADN entre tejido tumoral y tejido mamario sano, se estudiaron 11 perras enteras con tumores de mama. Ambos tipos de tejido fueron extraídos durante la cirugía, y se realizó la estadificación clínica y clasificación histopatológica de los tumores. A partir de muestras pareadas de tejido tumoral y tejido mamario sano de cada animal, se extrajo ADN y se determinaron los niveles de metilación genómica global mediante cuantificación relativa de 5-metil 2-desoxicitidina (5mdC) utilizando HPLC. Los resultados revelaron una hipometilación global del ADN en el tejido tumoral en comparación con el tejido mamario sano (P < 0.05). Esta diferencia fue más pronunciada en tumores de alto grado histopatológico, caracterizados por un comportamiento clínico agresivo y una elevada tasa de metástasis. Estos hallazgos subrayan la importancia de continuar con esta línea de investigación, ampliando el tamaño muestral. En el futuro, la metilación global del ADN podría utilizarse como un biomarcador pronóstico del cáncer de mama en perros.
Descargas
Citas
Zhou WM, Liu B, Shavandi A, Li L, Song H, Zhang JY. Methylation landscape: Targeting writer or eraser to discover anti-cancer drug. Front Pharmacol. [Internet]. 2021; 12:690057. doi: https://doi.org/qf7c DOI: https://doi.org/10.3389/fphar.2021.690057
Karsli-Ceppioglu S, Dagdemir A, Judes G, Ngollo M, Penault-Llorca F, Pajon A, Bignon YJ, Bernard-Gallon D. Epigenetic mechanisms of breast cancer: an update of the current knowledge. Epigenomics. [Internet]. 2014; 6(6):651-664. doi: https://doi.org/gms37n DOI: https://doi.org/10.2217/epi.14.59
Bártová E. Epigenetic and gene therapy in human and veterinary medicine. Environ. Epigenetics. [Internet]. 2024; 10(1):dvae006. doi: https://doi.org/qf7f DOI: https://doi.org/10.1093/eep/dvae006
Szczepanek J, Skorupa M, Jarkiewicz-Tretyn J, Cybulski C, Tretyn A. Harnessing Epigenetics for Breast Cancer Therapy: The Role of DNA Methylation, Histone Modifications, and MicroRNA. Int. J. Mol. Sci. [Internet]. 2023; 24(8):7235. doi: https://doi.org/qf7h DOI: https://doi.org/10.3390/ijms24087235
Brookes E, Shi Y. Diverse epigenetic mechanisms of human disease. Annu. Rev. Genet. [Internet]. 2014; 48:237-268. doi: https://doi.org/qf7j DOI: https://doi.org/10.1146/annurev-genet-120213-092518
Nishiyama A, Nakanishi M. Navigating the DNA methylation landscape of cancer. Trends Genet. [Internet]. 2021; 37(11):1012-1027. doi: https://doi.org/gm5thv DOI: https://doi.org/10.1016/j.tig.2021.05.002
Menezo Y, Silvestris E, Dale B, Elder K. Oxidative stress and alterations in DNA methylation: two sides of the same coin in reproduction. Reprod. Biomed. Online. [Internet]. 2016; 33(6):668-683. doi: https://doi.org/f9p3tc DOI: https://doi.org/10.1016/j.rbmo.2016.09.006
Pasculli B, Barbano R, Parrella P. Epigenetics of breast cancer: Biology and clinical implication in the era of precision medicine. Semin. Cancer Biol. [Internet]. 2018; 51:22-35. doi: https://doi.org/gd45tv DOI: https://doi.org/10.1016/j.semcancer.2018.01.007
Wu Y, Sarkissyan M, Vadgama JV. Epigenetics in Breast and Prostate Cancer. In: Verma, M. (eds). Cancer Epigenetics. Methods Mol. Biol. [Internet]. 2015; 1238:425–466. doi: https://doi.org/f6trhx DOI: https://doi.org/10.1007/978-1-4939-1804-1_23
Győrffy B, Bottai G, Fleischer T, Munkácsy G, Budczies J, Paladini L, Børresen-Dale A, Kristensen VN, Santarpia L. Aberrant DNA methylation impacts gene expression and prognosis in breast cancer subtypes. Int. J. Cancer. [Internet]. 2016; 138(1):87-97. doi: https://doi.org/gjjd2s DOI: https://doi.org/10.1002/ijc.29684
Berdasco M, Fraga M, Esteller M. Quantification of global DNA methylation by capillary electrophoresis and mass spectrometry. In: Tost, J. (eds) DNA Methylation. Methods Mol. Biol. [Internet]. 2009; 507:23–34. doi: https://doi.org/cjz5p9 DOI: https://doi.org/10.1007/978-1-59745-522-0_2
Cappetta M, Berdasco M, Hochmann J, Bonilla C, Sans M,Hidalgo PC, Artagaveytia N, Kittles R, Martínez M, Esteller M, Bertoni B. Effect of genetic ancestry on leukocyte global DNA methylation in cancer patients. BMC Cancer. [Internet]. 2015; 15:434. doi: https://doi.org/f7dp9f DOI: https://doi.org/10.1186/s12885-015-1461-0
Xavier P, Müller S, Fukumasu H. Epigenetic Mechanisms in Canine Cancer. Front. Oncol. [Internet]. 2020; 10:591843. doi: https://doi.org/qf7k DOI: https://doi.org/10.3389/fonc.2020.591843
Abdelmegeed S, Mohammed S. Canine mammary tumors as a model for human disease (Review). Oncol. Lett. [Internet] 2018; 15(6):8195–8205. doi: https://doi.org/mdsg DOI: https://doi.org/10.3892/ol.2018.8411
Jeong S, Lee K, Nam A, Cho J. Genome-wide methylation profiling in canine mammary tumor reveals miRNA candidates associated with human breast cancer. Cancers. [Internet] 2019; 11(10):1466. doi: https://doi.org/qf7m DOI: https://doi.org/10.3390/cancers11101466
Biondi L, Tedardi M, Gentile L, Chamas P, Dagli M. Quantification of global DNA methylation in canine mammary gland tumors via immunostaining of 5-methylcytosine: Histopathological and clinical correlations. Front. Vet. Sci. [Internet] 2021; 8:628241. doi: https://doi.org/qf7n DOI: https://doi.org/10.3389/fvets.2021.628241
Brandão Y, Toledo M, Chequin A, Cristo T, Sousa R, Ramos E, Klassen G. DNA methylation status of the estrogen receptor α gene in canine mammary tumors. Vet Pathol. [Internet] 2018; 55(4):510–516. doi: https://doi.org/qf7p DOI: https://doi.org/10.1177/0300985818763711
Beetch M, Harandi S, Yang T, Boycott C, Chen Y, Stefanskan B, Mohammed S. DNA methylation landscape of triple- negative ductal carcinoma in situ (DCIS) progressing to the invasive stage in canine breast cancer. Sci. Rep. [Internet] 2020; 10:2415. https://doi.org/qf7q DOI: https://doi.org/10.1038/s41598-020-59260-4
Cohen J. Statistical Power Analysis for the Behavioral Sciences. 2nd ed. New York University: Lawrence Erlbaum Associates. 1988.
Goldshmidt M, Peña L, Rasotto R, Zappulli, V. Classification and grading of canine mammary tumors. Vet. Pathol. [Internet] 2011; 48(1):117–131. doi: https://doi.org/cg36bk DOI: https://doi.org/10.1177/0300985810393258
Egenvall A, Bonnett B, Ohagen P, Olson P, Hedhammar A, von Euler H. Incidence of and survival after mammary tumors in a population of over 80,000 insured female dogs in Sweden from 1995 to 2002. Prevent. Vet. Med. [Internet]. 2005; 69(1-2):109–127. doi: https://doi.org/fw7k7z DOI: https://doi.org/10.1016/j.prevetmed.2005.01.014
Hillyar C, Rallis K, Varghese J. Advances in epigenetic cancer therapeutics. Cureus. [Internet] 2020; 12(11):e11725. doi: https://doi.org/gqsrqz DOI: https://doi.org/10.7759/cureus.11725